nnUNet 论文以及代码解析
nnUNet 论文:
nnU-Net: Self-adapting Framework for U-Net-Based Medical Image Segmentation
nnU-Net: a self-configuring method for deep learning-based biomedical image segmentation
代码:https://github.com/MIC-DKFZ/nnUNet
我会分成两部来写,上半部分是nnUNet论文里的内容,下半部分是nnUNet框架的使用。
在医学图像分割领域, nnUNet 总是一个绕不开的名字,每个新提出来的分割方法都要和 nnUNet 做对比,在某一个数据集上超越了 nnUNet,却在另一个不同的数据集上被 nnUNet 吊打,可以说到目前为止 nnUNet 依旧是医学图像分割的首选。
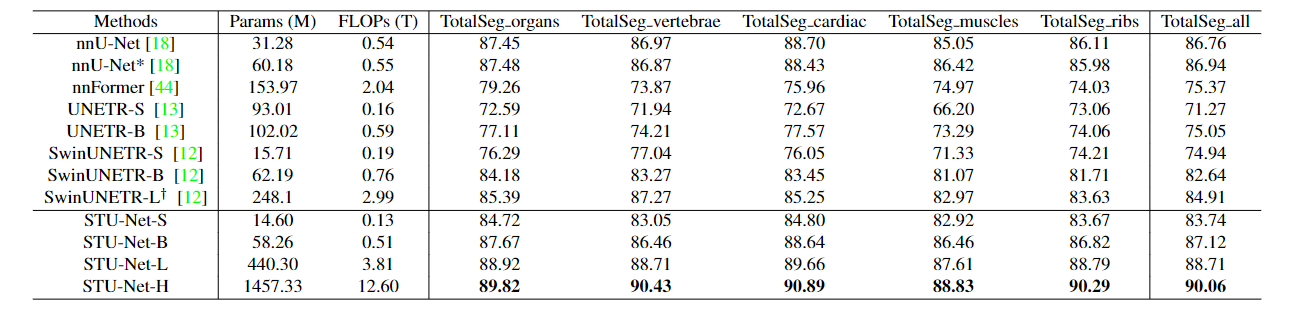 不同模型在 TotalSegmentator 数据集上的表现,结果来自STU-Net论文[1]
不同模型在 TotalSegmentator 数据集上的表现,结果来自STU-Net论文[1]
那么什么是 nnUNet ?nnUNet 是no-new-Net的缩写,指的是基于 2D 和 3D vanilla U-Nets 的鲁棒自适应框架。作者并没有加入一些花里胡哨的技巧,只是做了一些小改动,比如 ReLU 改成 Leaky ReLU 之类的。作者认为,与架构变化相比,方法配置的细节对性能的影响更大。
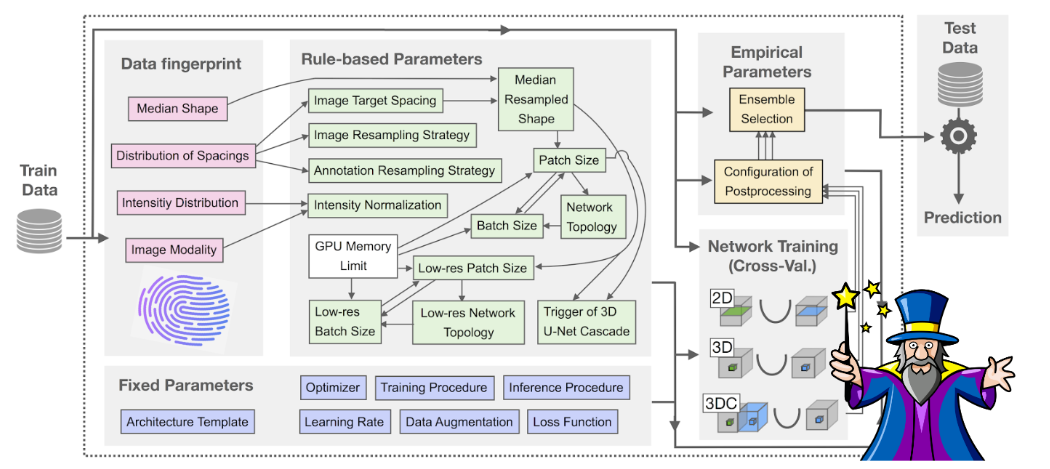
nnUNet 提出了一个可以自动配置的 pipline,包括了预处理、网络架构、训练和后处理,能够适应任何新的医学数据集。下面就介绍 nnUNet 的具体设置。
1. nnU-Net 的配置
nnU-Net 的自动配置基于将领域知识提炼为三个参数组:固定参数、基于规则的参数和经验参数。
下面解释配置细节。
数据集指纹
“数据集指纹”(‘dataset fingerprint’)是一种标准化的数据集表示法,包含图像大小、体素间距信息或类别比例等关键属性。
作为处理的第一步,nnU-Net 将提供的训练 case 裁剪到非零区域,从而提高了计算效率。根据裁剪后的训练数据,nnU-Net 创建了一个数据集指纹,其中包含所有相关参数和属性:裁剪前后的图像大小(即每个空间维度的体素数)、图像间距(即体素的物理大小)、模态(从 metadata 中读取)和所有图像的类别数,以及训练案例总数。此外,指纹还包括所有训练案例中前景区域强度值的平均值和标准差,以及 0.5 和 99.5 百分位数。
管道指纹
“管道指纹” (‘pipeline fingerprint’)被定义为方法设计时做出的全部选择。 nnU-Net 通过启发式规则自动推断,,并根据上述数据指纹和特定于项目的硬件约束进行操作。这些基于规则的参数由与数据无关的固定参数和在训练期间优化的经验参数进行补充.
1.1 固定参数
架构模板
nnU-Net 配置的所有 U-Net 架构均源自原始 U-Net 及其 3D 版本 。根据作者的假设,即配置良好的普通 U-Net 仍然难以超越,作者的 U-Net 配置都没有使用最近提出的架构变体,例如残差连接、密集连接、注意机制、squeeze-and-excitation 或空洞卷积。
作者只对原始架构进行了微小的更改。为了实现大 patch size,nnU-Net 中网络的batch size 较小。事实上,大多数 3D U-Net 配置的训练批量大小仅为 2。但 Batch normalization 在小 batch size下表现不佳。因此,作者对所有 U-Net 模型使用实例归一化 (instance normalization)。
此外,作者用 leaky ReLUs (负斜率,0.01)替换了ReLU。网络利用 deep supervision 训练:除了两个最低分辨率之外,解码器中还向所有分辨率添加了额外的辅助损失,从而允许将梯度更深入地注入到网络中,并促进网络中所有层的训练。
所有 U-Net 在编码器和解码器中都采用每个分辨率步骤两个 block 常见的配置,每个 block 由一个卷积组成,然后是实例归一化和 leaky ReLU。下采样由 strided convolution 实现,而上采样为卷积转置。作为性能和内存消耗之间的权衡,特征图的初始数量设置为 32,并在每次下采样(上采样)操作时加倍(减半)。为了限制最终模型的大小,3D 和 2D U-Net 的特征图数量分别被限制为320 和 512。
Training schedule
所有网络都经过 1,000 个 epoch 的训练,其中一个 epoch 被定义为超过 250 个 mini-batches的迭代。具有 Nesterov 动量 (μ = 0.99) 和初始学习率为 0.01 的随机梯度下降用于学习网络权重。学习率在整个训练过程中衰减,遵循“poly”学习率策略,$(1 − epoch/epoch_{max})^{0.9}$。损失函数是交叉熵和 Dice 损失 之和。对于每个深度监督输出,相应的下采样ound truth segmentation mask 用于损失计算。训练目标是所有分辨率下的损失 (L) 之和,各项的权重随着分辨率的每次降低而减半,并标准化为总和为 1。
Mini-batch 的样本是从随机训练案例中选择的。为了处理类别不平衡还实行了过采样: 66.7% 的样本来自所选训练案例中的随机选择,而 33.3% 保证包含前景类别之一。前景 patch 的数量强制最小值为 1 ,也就是一个随机 patch和一个前景patch,batch 大小为 2。在训练期间动态应用各种数据增强技术:旋转、缩放、高斯噪声、高斯模糊、亮度、对比度、低分辨率模拟、伽玛校正和镜像。
推理
使用滑动窗口方法预测图像,其中窗口大小等于训练期间使用的 patch 大小。相邻预测重叠了补丁大小的一半。越靠近窗口边界,分割的准确性就越低。为了抑制拼接伪影并减少靠近边界的位置的影响,应用高斯重要性加权,增加了 softmax 聚合中心体素的权重。通过沿所有轴进行镜像来实现测试时增强(TTA)。
1.2 基于规则的参数
强度标准化
nnU-Net 支持两种不同的图像强度归一化方案。除 CT 图像外,所有模式的默认设置都是 z scoring:在训练和推理期间,通过首先减去其平均值,然后除以其标准差,对每个图像进行独立归一化。如果裁剪导致平均尺寸减小 25% 或更多,则会创建中心非零体素的掩模,并且仅在该掩模内应用归一化,忽略周围的零体素。
对于 CT 图像,nnU-Net 采用不同的方案,因为强度值是定量的并反映组织的物理特性。因此,使用所有图像的全局归一化方案来保留此信息可能是有益的。为此,nnU-Net 使用前景体素的 0.5 和 99.5 百分位数对 HU 值进行剪切,并使用全局前景平均值和标准差对所有图像的进行标准化。
重采样
在某些数据集中,特别是在医学领域,体素间距(体素表示的物理空间)是异构的。为了应对这种异质性,nnU-Net 使用三阶样条、线性或最近邻插值以相同的目标间距对所有图像进行重新采样。图像数据的默认设置是三阶样条插值。对于各向异性图像(最大轴间距 ÷ 最小轴间距 > 3),平面内重采样采用三阶样条插值进行,平面外插值采用最近邻插值进行。在各向异性情况下以不同方式处理平面外轴可以抑制重采样伪影,因为切片之间的大轮廓变化更为常见。
通过将分割图转换为 one-hot 编码来重新采样。然后使用线性插值对每个通道进行插值,并通过 argmax 操作检索分割 mask。同样,各向异性情况会在低分辨率轴上使用 “最近邻 “进行插值。
Target spacing
Target spacing 是一个关键参数。较大的 spacing 会导致图像较小,从而损失细节;而较小的 spacing 则会导致图像太大,使网络无法积累足够的上下文信息,因为 patch 大小受限于给定的内存预算。
对于三维全分辨率 U-Net,nnU-Net 使用每个轴独立计算的 spacing 中值作为默认目标间距。对于各向异性的数据集,这一默认值可能会导致严重的插值伪影,或者由于训练数据的分辨率差异过大而导致大量信息丢失。因此,如果体素和间距各向异性(即最低间距轴与最高间距轴之比)均大于 3,则最低分辨率轴的目标间距将选择为训练案例中 spacing 的第 10 个百分位数。
对于二维 U-Net,nnU-Net 通常在分辨率最高的两个轴上运行。如果三个轴都是各向同性的,则使用两个拖尾轴进行切片提取。Target spacing 是训练样本的中位(每个轴独立计算)。对于基于切片的处理,无需沿平面外轴重新取样。
网络拓扑、 batch 大小和 patch 大小的调整
首先,作者列出所有不需要依据数据集进行的调整(例如网络结构为 “类 U-Net”),并优化它们的联合配置,以实现稳健的泛化。
其次,作者在 “数据集指纹”和 “管道指纹”之间建立了明确的依赖关系。依赖关系以相互依存的启发式规则形式建模,在应用时几乎可以立即执行。举例来说, batch size、patch size 和网络拓扑结构的相互依存配置基于以下三个原则:
- 较大的 batch size可以获得更准确的梯度估计,因此是可取的(在作者的领域中通常达不到最佳规模),但在实践中,任何大于 1 的 batch 都会影响训练的鲁棒性。
- 在训练过程中,较大的 patch size 会增加网络吸收的上下文信息,因此对性能至关重要。
- 网络的拓扑结构应该足够深,以保证有效感受野的大小至少与 patch 大小一样大,这样才不会丢弃上下文信息。
将这些知识提炼到方法设计中,就形成了以下启发式规则:
“将 patch size 初始化为图像形状中值,然后迭代减小补丁大小,同时相应调整网络拓扑结构(包括网络深度、沿每个轴进行池化操作的次数和位置、特征图大小和卷积核大小),直到在 GPU 内存限制的情况下,网络的 batch size 至少可以达到2”。
在适应过程中还考虑了图像 spacing:下采样操作可以被配置为,仅在 3D U-Net 中的特定轴和卷积核上操作,并且可以被配置为仅在某些图像平面上操作(伪 2D)。所有 U-Net 配置的网络拓扑都是根据重采样后的中值图像大小以及重采样图像的 Target spacing 来选择的。
初始化
重采样后,patch size 被初始化为中值图像形状。如果每个轴的 patch size 大小不能被 $2^{n_d}$ 整除(其中 nd 是下采样操作的数量),则会相应地进行填充。
架构拓扑
该架构是通过确定沿每个轴的下采样操作的数量来配置的,具体取决于 patch 大小和体素间距。执行下采样,直到进一步下采样将特征图尺寸减小到小于四个体素,或者特征图间距变得各向异性。下采样策略由体素间距决定;高分辨率轴被单独下采样,直到它们的分辨率在较低分辨率轴的两倍之内。随后,所有轴同时下采样。一旦触发相应的特征图约束,就会分别终止每个轴的下采样。 3D U-Net 和 2D U-Net 的默认卷积核大小分别为 3 × 3 × 3 和 3 × 3。如果轴之间存在初始分辨率差异(定义为大于 2 的间距比),则面外轴的内核大小将设置为 1,直到分辨率在 2 倍以内。请注意,所有轴的卷积核大小均保持为 3.
适应 GPU 内存预算
配置期间最大可能的patch size 受到 GPU 内存量的限制。由于patch size 在重采样后被初始化为中值图像形状,因此对于大多数数据集来说,它最初太大而无法放在 GPU。 nnU-Net 根据网络中特征图的大小来估计给定架构的内存消耗,并将其与已知内存消耗的参考值进行比较。然后在迭代过程中减小patch size ,同时在每个步骤中相应地更新架构配置,直到达到所需的预算。patch size 的减小始终应用于相对于数据的中值图像形状的最大轴。一次减小该轴 $2^{n_d}$ 个体素,其中 nd 是下采样操作的数量。
which means 换设备训练要重新配置一遍。
Batch size
最后一步是配置batch size。如果减少了 patch 大小,则 batch size设置为 2。否则,剩余的 GPU 内存空间将用于增加 batch size,直到完全使用 GPU。为了防止过拟合,batch size 受到限制,mini-batch 中的体素总数不超过所有训练案例的体素总数的5%。
3D U-Net 级联的配置
对下采样数据运行分割模型会增加与图像相关的 patch 大小,从而使网络能够积累更多上下文信息。这是以减少生成的分割中的细节为代价的,并且如果分割目标非常小或以其纹理为特征,也可能导致错误。因此,在具有无限 GPU 内存的假设场景中,通常倾向于以覆盖整个图像的块大小来以全分辨率训练模型。 3D U-Net 级联通过首先在下采样图像上运行 3D U-Net,然后训练第二个全分辨率 3D U-Net 以细化前者的分割图来近似这种方法。通过这种方式,“全局”低分辨率网络使用最大上下文信息来生成其分割输出,然后将其作为额外的输入通道来指导第二个“本地”U-Net。仅当 3D 全分辨率 U-Net 的 patch 大小覆盖小于中值图像形状的 12.5% 的数据集时,才会触发级联。如果是这种情况,下采样数据的目标间距和相关 3D 低分辨率 U-Net 的架构将在迭代过程中联合配置。
Target spacing 被初始化为全分辨率数据的 target spacing 。为了使 patch 大小覆盖输入图像的大部分,目标间距逐步增加 1%,同时在每一步中相应地更新架构配置,直到所得网络拓扑的 patch 大小超过当前中值图像的 25% 形状。如果当前spacing 是各向异性的,则仅增加较高分辨率轴的 spacing。级联的第二个 3D U-Net 的配置与独立 3D U-Net 的配置相同,其配置过程如上所述(除了第一个 U-Net 的上采样分割图连接到其输入)。
1.3 经验参数
U-Net 配置的集成和选择
nnU-Net 根据通过训练数据交叉验证计算出的平均前景 Dice 系数,自动确定用于推理的配置(ensemble)。所选模型可以是单个 U-Net(2D、3D 全分辨率、3D 低分辨率或级联的全分辨率 U-Net),也可以是这些配置中任意两个的组合。模型通过平均 softmax 概率来集成。
后处理
基于连接成分的后处理常用于医学图像分割。nnU-Net 遵循这一假设,自动评估抑制较小分量对交叉验证结果的影响。首先,所有前景类都被视为一个组件。如果除最大区域外,其他区域的抑制都能提高平均前景 Dice 系数,且不会降低任何类别的 Dice 系数,则选择此步骤作为第一个后处理步骤。最后,nnU-Net 基于此步骤的结果,并决定是否应对各个类别执行相同的过程。
2. nnUNet 应用
介绍了所有的细节,让我们按照运行的顺序把上面的各种配置捋一遍。
当在一个数据集上使用 nnUNet 时,首先 pipline 会收集数据集的信息,得到“数据集指纹”,保存为 dataset_fingerprint.json。
根据这些信息,进一步确定基于规则的参数。比如, 图像的 intensity distribution 和模态确定归一化方式,spacing 分布确定重采样策略以及 target spacing, target spacing 和 图像大小的中值确定 patch size,再结合GPU的内存确定 batch size 等。
基于参数的配置确定之后,nnUNet 会生成一个训练的 plan,名为 nnUNetPlans.json,包括确定的所有参数,在训练时 pipline会调用相关的 plan 处理数据、配置网络、进行训练等。
固定参数包括:网络结构模板,数据增强,loss,learning rate,optimizer, 训练流程等。
3. 在自己的数据集训练上使用 nnUNet
这里写的是关于 nnUNetv2 的使用。
首先需要自己安装好 torch, 如果只是想用预训练好的模型进行推理,可以直接使用pip install nnunetv2。如果需要代码,就 git clone 到本地,注意第三行的那个点:
1
2
3
git clone https://github.com/MIC-DKFZ/nnUNet.git
cd nnUNet
pip install -e .
3.1 数据准备
首先我们需要把自己的数据按照 nnUNet 需要的数据格式命名和储存。
图像文件的命名格式为:{CASE_IDENTIFIER}_{XXXX}.{FILE_ENDING}
其中,CASE_IDENTIFIER 是每一个 case 的唯一 id,比如病人的编号。XXXX 是 4 位数字的模态/通道标识符(对于每个模态/通道应该是唯一的,例如,“0000”表示 T1,“0001”表示 T2,只有一个模态就是“0000”)。FILE_ENDING 是图像格式使用的文件扩展名(.png、.nii.gz、…)。
分割文件的命名格式为: {CASE_IDENTIFER}.{FILE_ENDING}
注意到分割文件里没有通道标识符,意味着每个 case 的所有模态 or 通道共享一个分割文件,如果你的分割文件不一样,最好是把它们当做两个 case。
分割必须与其相应的图像共享相同的几何形状,每个像素值都是整数,代表一个语义类别。背景必须为 0。如果没有背景,也不要将标签 0 用于其他类别,语义类别的整数值必须是连续的(0、1、2、3…)。
在给所有文件命名好之后,我们需要将他们按照 nnUNet 需要的文件夹里。在喜欢的路径下新建一个文件夹 nnUNet_raw, 将自己的数据集命名为 Dataset{XXX}_{NAME},需要注意的是 XXX 为 001 到 010 的是数据集 MSD 的编号,所以需要避开它们。
在 nnUNet_raw 下面新建一个文件夹名为 Dataset{XXX}_{NAME}, 在数据集文件夹下新建几个文件夹和一个文件,包括:
-imagesTr 里放所有命名好的训练图像。 nnU-Net 将使用这些数据进行管道配置、交叉验证训练、后处理和最佳集合。
- imagesTs(可选)包含属于测试用例的图像。 nnU-Net 不使用它们,放着方便。
- labelsTr 所有训练集的 groundtruth 分割图像。
- dataset.json 包含数据集的元数据。
以 BRTAS 数据集为例, 最终的文件夹结构是这样的:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
nnUNet_raw/Dataset001_BrainTumour/
├── dataset.json
├── imagesTr
│ ├── BRATS_001_0000.nii.gz
│ ├── BRATS_001_0001.nii.gz
│ ├── BRATS_001_0002.nii.gz
│ ├── BRATS_001_0003.nii.gz
│ ├── BRATS_002_0000.nii.gz
│ ├── BRATS_002_0001.nii.gz
│ ├── BRATS_002_0002.nii.gz
│ ├── BRATS_002_0003.nii.gz
│ ├── ...
├── imagesTs
│ ├── BRATS_485_0000.nii.gz
│ ├── BRATS_485_0001.nii.gz
│ ├── BRATS_485_0002.nii.gz
│ ├── BRATS_485_0003.nii.gz
│ ├── BRATS_486_0000.nii.gz
│ ├── BRATS_486_0001.nii.gz
│ ├── BRATS_486_0002.nii.gz
│ ├── BRATS_486_0003.nii.gz
│ ├── ...
└── labelsTr
├── BRATS_001.nii.gz
├── BRATS_002.nii.gz
├── ...
nnUNetv2 支持 png 以及许多医学图像常见的格式包括:
- NaturalImage2DIO:.png、.bmp、.tif
- NibabelIO:.nii.gz、.nrrd、.mha
- NibabelIOWithReorient:.nii.gz、.nrrd、.mha。请注意 nibabel会更改图像方向为 RAS(RAS指的是方向坐标 from left to Right; posterior to Anterior; and inferior to Superior)。
- SimpleITKIO:.nii.gz、.nrrd、.mha
- Tiff3DIO:.tif、.tiff。 3D tif 图像!由于 TIF 没有存储间距信息的标准化方法,因此 nnU-Net 希望每个 TIF 文件都附带一个包含此信息的同名 .json 文件。
dataset.json 包含 nnU-Net 训练所需的元数据, 内容如下:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
{
"channel_names": { # 模态/通道名, nnUNet 其实只关心它是不是CT,因为CT 的归一化方式不一样。
"0": "T1",
"1": "T2"
},
"labels": { # 这里的分割图上的类别名
"background": 0,
"PZ": 1,
"TZ": 2
},
"numTraining": 32,
"file_ending": ".nii.gz"
"overwrite_image_reader_writer": "SimpleITKIO" # optional! 可选!如果不指明,nnU-Net 将自动确定。
}
}
可以在 nnUNet/nnunetv2/dataset_conversion/generate_dataset_json.py 中调用函数生成需要的 dataset.json 文件。
3.2
整理完数据后就可以进入轻松愉快的自动配置环节了。
打开命令行,输入下面的命令进行数据预处理以及生成配置文件:
1
nnUNetv2_plan_and_preprocess -d DATASET_ID --verify_dataset_integrity
其中 DATASET_ID 是数据集 ID(废话)。每当第一次运行此命令时,作者建议使用 –verify_dataset_integrity 检查一些最常见的错误。
运行 nnUNetv2_plan_and_preprocess -h 可以获得更多信息, 当然也可以直接找到 nnUNet/nnunetv2/experiment_planning/plan_and_preprocess_entrypoints.py 看看它代码究竟怎么写的。
如果没有什么问题, 等到这上面这行命令运行结束,就可以开始训练了。
3.3 训练
在 1.3 中我们提到了 uuNet 有四种配置:2D、3D 全分辨率、3D 低分辨率和级联的全分辨率 U-Net, 对应的配置名分别为 2d, 3d_fullres, 3d_lowres, 3d_cascade_fullres。需要注意根据数据集 3d_lowres 配置可能被跳过。
训练命令如下:
1
nnUNetv2_train DATASET_NAME_OR_ID UNET_CONFIGURATION FOLD
其中,DATASET_NAME_OR_ID 是要训练的数据集 id 或者名字(建议别写名字,好麻烦)。
UNET_CONFIGURATION 是 2d, 3d_fullres, 3d_lowres, 3d_cascade_fullres 中的一个, 请注意 3d_cascade_fullres 需要在完成5折的 3d_lowres 之后进行,3d_lowres 训练完成后会生成一个 predicted_next_stage 文件夹,这是运行 3d_cascade_fullres 必需的。
FOLD 是第几折,从 0 到 5 的整数。
接下来是几份常用的参数:
- –c, 继续训练
- -tr TrainerName,用指定的 trainer 训练,比如 nnUNetTrainer_5epochs,即改变训练 epoch 为 5, trainer 的各种变体在
nnunetv2/training/nnUNetTrainer/variants。 - –val,只验证,默认为 checkpoint_final.pth
- –val_best,使用 checkpoint_best.pth
如果你想要在多个 GPU 上训练的话,建议不要用 -num_gpus, 而是:
1
2
3
4
5
6
7
CUDA_VISIBLE_DEVICES=0 nnUNetv2_train DATASET_NAME_OR_ID 2d 0 [--npz] & # train on GPU 0
CUDA_VISIBLE_DEVICES=1 nnUNetv2_train DATASET_NAME_OR_ID 2d 1 [--npz] & # train on GPU 1
CUDA_VISIBLE_DEVICES=2 nnUNetv2_train DATASET_NAME_OR_ID 2d 2 [--npz] & # train on GPU 2
CUDA_VISIBLE_DEVICES=3 nnUNetv2_train DATASET_NAME_OR_ID 2d 3 [--npz] & # train on GPU 3
CUDA_VISIBLE_DEVICES=4 nnUNetv2_train DATASET_NAME_OR_ID 2d 4 [--npz] & # train on GPU 4
...
wait
个人感觉是使用多个 GPU 训练的时候反而 CPU 和 IO 会成为瓶颈,导致多卡训练时每个卡上的训练时间比只用单卡长一些,看个人设备吧。
一旦训练完成(完全交叉验证),就可以让 nnU-Net 自动确定最佳配置:
1
nnUNetv2_find_best_configuration DATASET_NAME_OR_ID -c CONFIGURATIONS
CONFIGURATIONS 是想要搜索的配置列表。默认情况下,ensemble 是启用的,这意味着 nnU-Net 将生成所有可能的集成组合(每个集成有 2 个配置)。这个命令需要包含验证集预测概率的 .npz 文件(使用 nnUNetv2_train 的时候加上 –npz )。
3.4 预测
1
nnUNetv2_predict -i INPUT_FOLDER -o OUTPUT_FOLDER -d DATASET_NAME_OR_ID -c CONFIGURATION --save_probabilities
–save_probabilities 将预测概率与预测的 mask 一起保存。
-f 指定使用某一折的模型,或者用全部的 5折 –f all。
关于更多的信息可以看 nnUNet 的文档。
参考:
- [1] STU-Net: Scalable and Transferable Medical Image Segmentation Models Empowered by Large-Scale Supervised Pre-training